 期刊:Science China-Life Sciences
期刊:Science China-Life Sciences
影响因子:8.0
常规多组学技术,目前已被广泛应用于疾病发展、组织发育等研究领域。在单细胞水平,从相同或相似来源的不同细胞中获取多种组学信息,通过单细胞转录组鉴定细胞类型后关联到其他相似细胞的不同组学层面,以发现新的细胞亚群和新的生物学机制。测序技术的高速发展使得在单细胞分辨率下检测同一细胞中的DNA、mRNA、表观基因组和蛋白质组等信息成为可能。
为了整合单细胞多组学数据,目前已经开发了多种生物信息学算法。在本章中,作者回顾了:
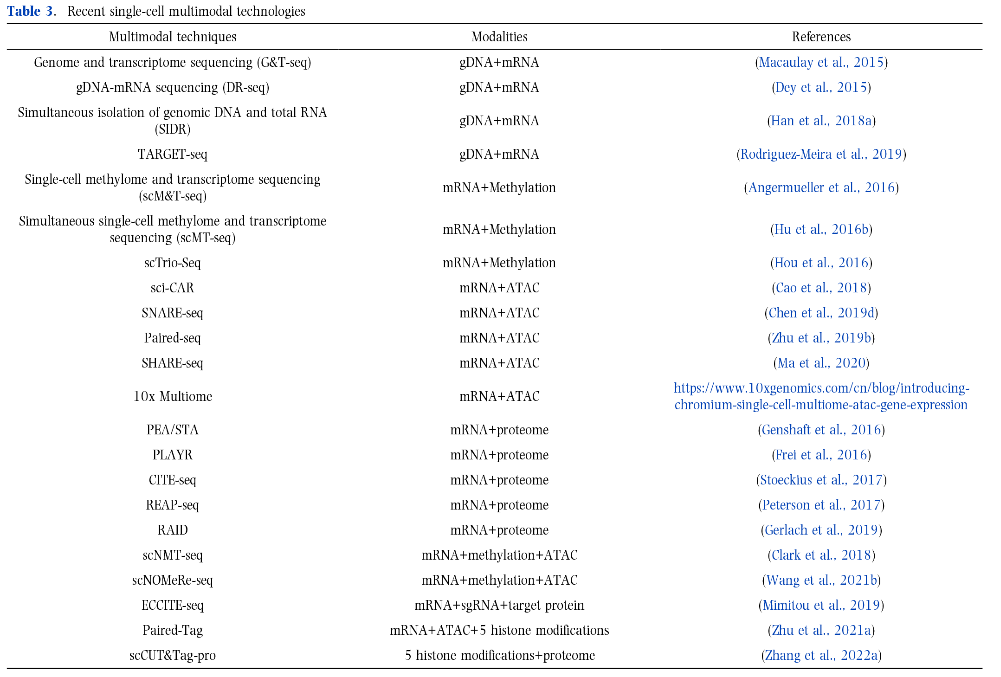
(1)捕获同一细胞的单细胞多组学技术的最新发展,主要集中在以转录组为核心的多组学技术(表3);
(2)多组学数据整合分析方法和工具的最新进展,包括针对未配对多组学数据集和配对多组学数据集的应用和算法;
(3)总结单细胞多组学整合分析基准研究中数据整合方法的性能。
单细胞多组学测序
通过单细胞多组学技术,可以分析同一细胞的多种类型的分子。近年来,人们开发了多种单细胞测序方法,用于捕获基因组DNA(gDNA)、转录组、蛋白质组和表观基因组。单细胞多组学测序技术工作流程的主要步骤如图13所示。
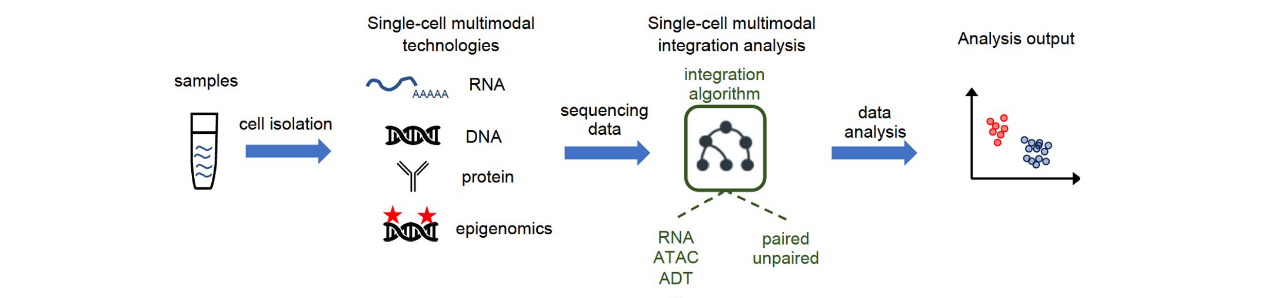
图13 单细胞多组学测序技术的主要实验流程
1. 转录组+gDNA
目前已有多种技术可以同时测量单个细胞中的mRNA和gDNA。G&T-seq(Genome and transcriptome sequencing)应用流式细胞术分离细胞,使用beads分离细胞中的mRNA和gDNA。DR-seq(gDNA-mRNA sequencing)通过移液管分选细胞,然后分离和扩增标记的gDNA和mRNA。SIDR技术 (Simultaneous isolation of genomic DNA and total RNA)使用微孔板分选细胞,并通过低渗胞饮法分离细胞核和细胞质。TARGET-seq技术优化了FACS的细胞分离和逆转录聚合酶链反应(RT-PCR)扩增步骤,提高了细胞通量。
2. 转录组+表观基因组
亚硫酸氢盐(BS)处理可以转化发生甲基化和未发生甲基化的DNA CG位点,并通过PCR和二代测序在单核苷酸分辨率上分析DNA甲基化。基于该原理,目前已经开发了多种单细胞甲基化测序技术,用于检测单细胞水平上的甲基化修饰水平,包括scRRBS、scWGBS、snmC-seq和sci-MET。同样的,目前已开发多种单细胞多组学技术,捕获同一细胞中的mRNA和gDNA的甲基化。首先,scM&T -seq(single-cell methylome and transcriptome sequencing)使用与G&T-seq类似的方法,利用流式分离细胞,基于beads分离细胞的mRNA和gDNA,然后进行亚硫酸氢盐处理。scMT-seq(simultaneous single-cell methylome and transcriptome sequencing)使用微量移液法从单细胞裂解物中分离细胞核,并通过scRRBS和改良的Smart-seq2流程分别生成DNA甲基化和转录组数据。此外,scTrio-seq可以分析同一细胞中的基因组CNVs、DNA甲基化和转录组,其中基因组CNVs可以通过大量RRBS数据从scRRBS中计算推断出来。
多种基于二代测序的技术,如ChIPseq、Dnase -seq、ATAC-seq,用于研究表观基因组图谱,如染色质结构和组蛋白修饰。另一种类似的方法,NOMe-Seq (Nucleosome Occupancy and Methylome Sequencing)可以使用外源性M. CviPI GpC甲基转移酶标记开放的基因组区域,同时测定核小体占位和甲基化水平。在这些方法的基础上,还开发了许多新的技术来测量染色质可及性、DNA甲基化或单细胞分辨率下染色质可及位点的组蛋白修饰,如scDNase-seq、sci-ATAC seq、scATAC-seq、scMNase-seq、scChIP-seq等,可以检测H3K4me3和H3K4me2修饰。
基于这些技术,开发了多种联合检测染色质可及性和转录组的单细胞多组学高通量方法。Cao等人(2018)开发了sci-CAR,是第一个可以同时分析同一细胞中mRNA和ATAC的技术。Sci-CAR对每个细胞进行组合索引,通过FACs分离细胞,裂解后进行扩增测序。Cao等人(2018)将sci-CAR技术应用于人类和小鼠细胞系混合物以及小鼠肾组织,并从联合分析数据集中确定了顺式调控网络。然而,由于scATAC数据模式的高稀疏性和scRNA模式测序深度有限,在sci-CAR数据集中只能重现少数其他scRNA 和 scATAC 测序中的差异可及位点和差异表达基因。Chen等(2019d)开发了基于液滴的SNARE-seq(single-nucleus chromatin accessibility and mRNA expression sequencing),增强了scRNA和scATAC的测序覆盖范围,并改善了sci-CAR技术的覆盖限制。SNARE-seq使用夹板寡核苷酸,其序列与 ATAC 转座酶插入的接头序列(5' 端)和 mRNA polyA序列互补,可以捕获两个组学数据。与 sci-CAR 相比,SNARE-seq 在小鼠脑数据集和成人脑数据集中检测到的染色质可及位点是 sciCAR 组织数据集的4-5倍,并通过细胞组合索引策略提高了检测通量。Zhu等(2019b)进一步完善了该方案,开发了Paired-seq,通过测序同时检测单个细胞中的RNA表达和DNA可及性。其主要是采用基于连接的组合索引策略,通过 Tn5 转座酶切割开放染色质片段,通过RNA反转录得到cDNA 分子。使用Paired-seq技术检测小鼠胚胎大脑皮层组织,并与ENCODE小鼠胚胎大脑皮层组织数据集进行整合分析,重建细胞轨迹,并从双组学数据集中恢复了顺式调控网络。Ma等人(2020)开发了SHARE-seq技术(simultaneous high-throughput ATAC and RNA expression with sequencing),该方法使用多轮杂交阻断来联合标记同一单细胞中的mRNA和染色质片段。与sciCAR、SNARE-seq和Paired-seq相比,SHARE-seq在超过30,000个细胞的更大文库上具有更高的可扩展性,并且在每个细胞中检测到更多的基因和ATAC peak时具有更高的灵敏度。基于更高的数据质量,Ma等人给出了一个新的计算策略DORCs(domains of regulatory chromatin,调控染色质域),而不是单个峰来分析染色质可及性和基因表达之间的调控图谱,DORCs在细胞谱系选择和细胞命运决定中优于基因表达,可以更好的预测细胞命运。最近,10x Genomics 开发了10x Multiome,一个用于单细胞中scRNA和scATAC联合检测的商业服务平台,这将加速scRNA和scATAC多组学技术在更多生物学和临床研究中的应用。
3. 转录组+蛋白组
除了针对DNA和RNA的单细胞多组学,还开发了多种可以同时测量同一细胞中RNA和蛋白质的单细胞技术。PEA/STA技术(proximity extension assay/specific RNA target amplification)使用逆转录酶作为DNA聚合酶,用于RNA的反转录和38种蛋白PEA中DNA寡核苷酸的延伸,以使cDNA合成和PEA能够同时在 Fluidigm C1 TM 系统中进行。PLAYR(proximity ligation assay for RNA)技术是一种使用流式细胞术和质谱流式细胞技术进行多重转录物定量的方法,与标准抗体染色兼容。PLAYR允许同时测量40多种mRNA和蛋白质,并能够在单细胞水平上表征转录和翻译之间的相互作用。除了蛋白-RNA联合检测外,还开发了两种靶向表面蛋白和mRNA的方法:CITE-seq和REAP-seq,这两种技术使用寡核苷酸标记的抗体检测mRNA和细胞表面蛋白,并通过基于液滴的单细胞测序方法实现单细胞水平的多组学分析,极大地提高了转录组的通量。例如,REAP-seq可以用82种抗体定量蛋白质,并在一次实验中检测超过20,000个基因。RAID(single-cell RNA and immunodetection)可以检测细胞内蛋白和磷酸化蛋白以及mRNA。RAID用连接RNA条形码的抗体对细胞内靶蛋白进行免疫标记,然后将蛋白转化为RNA。
4. 捕获两种以上组学的技术
基于上面讨论的方法,还开发了多种方法检测同一个细胞中两个以上的组学。scNMT-seq(Single-cell nucleosome, methylation and transcription sequencing)将scM&T-seq和NOMe-seq相结合,用于测量同一细胞中的核小体、转录组和DNA甲基化信息。scNOMeRe-seq,能够在同一个细胞中分析全基因组染色质可及性、DNA甲基化和转录组。基于CITE-seq,开发了ECCITE-seq(Expanded CRISPR-compatible Cellular Indexing of Transcriptomes and Epitopes by sequencing),可以同时检测单细胞中的蛋白、转录组、克隆型和CRISPR信息。通过整合pair -seq与CUT&Tag技术,开发了Paired-Tag技术,可以同时分析同一细胞中scRNA,scATAC和五种组蛋白修饰。scCUT&Tag-pro技术,也是将CUT&Tag与CITE-seq结合,通过CUT&Tag文库捕获5个组蛋白修饰,通过抗体衍生蛋白标签文库捕获蛋白质。
单细胞多组学技术的发展为多视角、多维度、高分辨率的揭示分子机制提供了丰富的数据资源。然而,由于数据维数高、数据稀疏度高以及多组学数据集和技术之间变量复杂等问题,难以对多组学数据集进行正确的整合分析。目前越来越多的算法被开发出来,本文从以下两个角度介绍了多组学整合分析算法和相关研究,包括(1)多组学整合工具的类别,其中介绍了最近发表的单细胞多组学整合工具,并按照不同的分类标准进行分类;(2)最近发表的工具的基准研究。
1. 多组学整合工具的分类
基于之前的综述论文,可以应用几个标准对多组学整合工具进行分类。首先,基于数据集的共同特征(称为锚点)的选择,多组学整合分析可以分为四种主要的整合策略,包括以所有基因组特征(如基因)为锚点的水平整合,以所有共同细胞为锚点的垂直整合,无共享特征的对角线整合以及以部分共享细胞和部分共享基因组特征为锚点的马赛克整合。具有相似方法学的工具也可能被开发为不同类型。例如,对于基于非负矩阵分解 (NMF) 的算法,iNMF被设计为垂直整合工具,coupledNMF被设计为对角线整合工具,UINMF被设计为马赛克整合工具。
其次,多组学整合工具也可以根据多组学技术的类型分类,包括“配对”和“非配对”整合工具。配对的多组学整合工具是专门为同时从同一细胞捕获和测序的多组学数据集而设计的。配对整合工具通常采用垂直整合和镶嵌整合策略,因为配对数据集在不同组学之间共享全部或部分细胞。非配对整合工具旨在整合来自不同细胞的多种单细胞组学数据,因为一种组学的细胞无法从其他组学中找到匹配的细胞。由于细胞和基因的差异,对角线整合通常用于非成对数据整合。针对配对或非配对多组学数据集整合,开发了一些工具。例如,Seurat v3是为未配对的多组学数据集设计的,而更新版本Seurat v4是专门为配对的多组学数据集设计的。一些整合工具可以同时应用于成对和非成对的多组学数据集。
第三,基于方法论,多组学整合工具可以分为:数学矩阵分解方法、流形对齐方法、基于网络的方法和深度学习方法。深度学习整合工具可以根据深度模型的基础结构进一步分类,包括自编码器(AE)、生成对抗网络(GAN)、GNN及其扩展结构,如变分自编码器(VAE)。选择哪种方法很大程度上取决于多组学数据集的类型和整合目的。对于未配对的多组学数据集,流形对齐方法可以首先将多组学数据集的不同特征降低到同一维度的潜在嵌入/流形中,然后通过相同的流形整合异构组学。同样,矩阵分解方法可以通过将不匹配的基因或细胞矩阵分解为相同维度的矩阵以应用于不同的整合任务,与简单的降维方法以及流形对齐相比,信息损失更少。使用GAN, VAE和transformer的深度学习工具可以从相同细胞或共享细胞的不同组学中学习到共同的潜在嵌入信息,然后填补缺失的细胞和基因,如GNN模型用于学习不同类型的特征(如scRNA中的基因和scATAC中的peak等)之间的关系,并推断多组学数据中的生物网络。
第四,根据特定的组学对多组学整合工具进行分类。针对特定的多组学数据集整合,设计了一些多组学整合算法;例如,CiteFuse是为CITE-seq分析而设计的,SCIM是为scRNA和CyTOF整合而设计的,而scMVP是为配对的scRNA和scATAC数据集整合而开发的。此外,除了这些仅限于特定组学数据类型的工具外,还有一些算法,如LIGER是为一般性整合任务而设计的,不受整合组学类型的限制。
多组学整合工具还可以按Python、R等主要编码语言以及跨组学翻译等特殊整合应用程序进行分类。所有多组学整合工具及其类别总结在补充材料中。
2. 单细胞多组学整合工具的基准研究
尽管针对多组学单细胞分析已经发表了大量的整合算法,但仍然很难找到最优解。为了解决这个问题,最近有多项基准研究,试图从数据用户的角度对候选整合算法的性能进行全面而客观的评估。
Luecken等人(2022)对单细胞整合工具进行了基准分析,用于图谱级数据整合任务,并开发了一个对单细胞整合工具进行客观、全面和可重复评估的基准流程。该研究包括几个未配对的多组学单细胞整合工具;然而,该研究只针对来自相同组学的不同数据集的整合任务,如不同scRNA数据集的整合或不同scATAC数据集的整合,而没有提供评估配对或未配对的scRNA和scATAC数据的跨组学整合。尽管如此,本研究仍然为单细胞图谱整合评估提供了一个稳定、全面、高度可扩展的基准框架。
最近,为了更好地应对单细胞多组学整合分析的数据稀疏性、技术和生物可变性以及高维度带来的分析挑战,NeuraIPS2021组织了关于整合单细胞多组学数据三个主要任务的在线竞赛,包括(1)从一种组学预测另一种组学,(2)不同组学之间的细胞匹配,(3)共同学习细胞身份的表征。其中,第二项任务是针对非配对多组学整合工具设计的,第三项任务是针对配对多组学整合工具设计的。此外,竞赛还生成了第一个单细胞多组学基准数据集,包括一个CITE-seq数据集,其中包含90000个细胞,用于scRNA和蛋白质整合任务,以及一个10x Multiome数据集,包含70000个细胞,用于scRNA和scATAC整合任务。在三个任务中,GLUE包中的半监督模式匹配函数CLUE(cross-linked universal embedding)算法在第二个组学匹配任务中获得一等奖并获得所有类别的冠军,显示了在未配对整合工具中交叉组学匹配的最佳性能。然而,由于竞赛只评估在线提交者的算法,大多数已发表的单细胞多组学整合工具不在评估范围内。
为了进一步比较已发表的单细胞多组学整合工具与深度学习框架,Brombacher等人(2022)首先使用深度学习模型回顾了18个最近发表的多组学整合工具,然后使用来自NeuraIPS2021的CITE-seq数据集和10x Multiome数据集对选定的工具进行了第一次全面的基准研究。在生物特征保存任务中,Cobolt算法在CITE-seq和10x Multiome数据集上的性能都是基准算法中最好的。只有当细胞数较大时,scMVP在10x Multiome数据集上的性能优于Cobolt。技术影响去除任务中,SCALEX在CITE-seq数据集上表现出的最佳性能,而scMVP在10x Multiome数据集上表现出最佳性能。
Sun F, Li H, Sun D, et al. Single-cell omics: experimental workflow, data analyses and applications. Sci China Life Sci. 2025;68(1):5-102. doi:10.1007/s11427-023-2561-0
